Oncogene - Mutant p53 übt einen dominant negativen Effekt durch die Verhinderung Wildtyp-p53 aus der Bindung an
Mutant p53 übt eine dominant negative Wirkung durch Verhinderung Wildtyp p53 aus an den Promotor seiner Zielgene bindenden
Amy Willis 1. Eun Joo Jung 1. Therese Wakefield 1 und Xinbin Chen 1
1 Abteilung für Zellbiologie und UAB Comprehensive Cancer Center, The University of Alabama in Birmingham, Birmingham, AL 35294-0005, USA
p53, p53-Mutante, Zellzyklusarrest, Transkriptions
Einführung
Die Expression von mutierten und Wildtyp-p53 im dualen System induzierbaren
Etablierung von stabilen Zelllinien induzierbar exprimieren mutiertes p53 Wildtyp-p53 oder beide gleichzeitig Proteine. Unter Verwendung des Dual-induzierbare System wurden Zellinien etabliert, die induzierbar Wildtyp-p53 und p53 eine der Hot-Spot-Mutanten exprimieren kann: R175H (a), R248W (b) oder R273H (c). Die Zellen wurden induziert weder Protein (Spur 1), p53-Mutante alleine (Spur 2), Wildtyp-p53 allein (Spur 3) oder beiden Proteine gleichzeitig (Bahn 4) für 24 h zum Ausdruck bringen. Zellextrakte wurden gesammelt und die Proteine durch SDS-PAGE aufgetrennt. p53-Expression wurde durch Sondieren mit p53-Antikörper PAb1801 und Pab421 nachgewiesen. p21-Expression wurde mit dem polyklonalen Antikörper p21 nachgewiesen. Die Membran wurde mit Actin als Ladekontrolle sondiert
Ganzfigurig und Legende (113K)
Mutant und Wildtyp-p53 Interact physikalisch und koexprimiert werden, wenn beide in dem dualen induzierbaren System exprimiert werden,
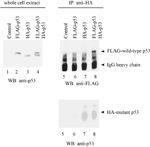
Mutant und Wildtyp-p53 interagieren physisch in vivo. Zellextrakte wurden aus Zellen (Zelllinie, hergestellt # 035; 208) nicht-induzierten (Bahn 1 und 5), induzierte HA-markierte p53-Mutante (R273H) (Spuren 3 und 7), FLAG-markierten Wildtyp-p53 (Bahnen 2 und 6) auszudrücken oder beide (Spuren 4 und 8). Für Spuren 1-4 wurde die Western-Blot mit anti-p53-Antikörper sondiert. Für Spuren 5-8 wurden die Zelllysate mit anti-HA-Antikörper (Spuren 5-8), und die Proteine in den Immunpräzipitaten immunpräzipitiert wurden dann mit anti-FLAG-Antikörper (rechts auf der Oberseite) oder anti-p53-Antikörper (rechte Tafel detektiert auf der Unterseite)
Ganzfigurig und Legende (81K)
Obwohl Mutante und Expression Wildtyp-p53 in unseren stabilen Zelllinien ausgewählt wurde, wollten wir sicherstellen, dass diese Proteine in derselben Zelle exprimiert wurden. unter Verwendung von Immunfärbung von HA-markiertem p53-Mutante (R273H) und FLAG-markierten Wildtyp-p53 Dies wurde erreicht. Wie in Abbildung 3 gezeigt mutierten p53 wurde durch polyklonale anti-HA-Antikörper (rot) und Wildtyp-p53 durch den monoklonalen anti-FLAG-Antikörper (grün) in der gleichen Zelle erkannt wird, wenn beide Proteine induziert wurden. Wenn diese Bilder zusammengeführt wurden, wurde das Fluoreszenzsignal verändert, was darauf hindeutet, dass mutierte und Wildtyp-p53 in der gleichen Kammer von Zellkern lokalisiert waren. Daher ist die verminderte Induktion von p21 wahrscheinlich aufgrund der direkten dominant negativen Wirkung des mutierten p53 auf Wildtyp-p53.
Mutant und Wildtyp-p53 in der gleichen Zelle exprimiert wird. Die Zellen wurden in vier-Well-Kammerobjektträger ausgesät, für 24 h in geeigneten Induktionsmedium induziert und für die Mutante (anti-HA) und Wildtyp (anti-Flag) p53 gefärbt. Kernfärbung ist blau, mutiertes p53 Fleck ist rot, und Wildtyp-p53-Färbung ist grün
Ganzfigurig und Legende (75K)
Mutant p53 hemmt die Fähigkeit von Wildtyp-p53 seine Zielgene zu trans
Ganzfigurig und Legende (130K)
Mutant p53 hemmt die Fähigkeit von Wildtyp-p53 zu p53 ansprechenden Elemente in seinen Zielgen zu binden,

Ganzfigurig und Legende (83K)
Mutant p53 reduziert die Fähigkeit von Wildtyp-p53 Zellproliferation zu unterdrücken
Mutant p53 hemmt die Fähigkeit von Wildtyp-p53 Zellproliferation zu unterdrücken. Die Zellen wurden induziert weder Protein, p53-Mutante allein, Wildtyp-p53 allein oder beide gleichzeitig zum Ausdruck bringen. Wachstumsraten wurden über 5 Tage ausgewertet
Ganzfigurig und Legende (77K)
Dominant-negative mutierte p53 stört Induktion von Zellzyklusarrest von Wildtyp-p53

Mutant p53 hemmt die Fähigkeit von Wildtyp-p53 Zellzyklusarrest zu induzieren. Die Zellen wurden induziert weder Protein (Kontrolle), mutanten p53 allein, Wildtyp-p53 allein oder beiden Proteine gleichzeitig (Mutante + Wildtyp) auszudrücken. DNA-Histogramm-Analyse wurde verwendet, um den Prozentsatz an Zellen in jeder Phase des Zellzyklus zu bestimmen, wie durch Propidiumiodid-Färbung von DNA nachgewiesen
Ganzfigurig und Legende (183K)
Diskussion
Um den Mechanismus der dominant negativen Aktivität zu bestimmen, haben wir Chip durchgeführt, zu untersuchen, ob p53-Mutante verhindert, dass das Wildtyp-Protein von sequenzspezifischen auf den Promotor seiner Zielgen binden. Dieses Verfahren ist vorteilhaft, weil es ermöglicht Erforschung der DNA in vivo zu binden. Das duale System ermöglicht induzierbares Vergleich dieses in Zellen bindenden p53-Wildtyp allein auf Zellen, die Wildtyp-p53 in der Anwesenheit eines mutierten p53 exprimieren. So haben unsere Daten überzeugenden Beweise vorausgesetzt, dass die dominant-negative Aktivität von mutiertem p53 ist durch die Hemmung von Wildtyp-p53 zu p53 ansprechendem Elemente zu binden, und anschließend, deren Transkriptionsaktivität.
Materialen und Methoden
Einrichtung eines dualen induzierbaren Systems und stabile Zelllinien
Immunpräzipitation und Western-Analysen
Wachstumsrate Analysen
Die Zellen wurden bei etwa 60 000 Zellen / 60-mm-Platte ausgesät. Die Zellen wurden induziert weder Protein, mutanten p53 allein, Wildtyp-p53 allein oder beiden Proteine gleichzeitig zu exprimieren. Das Medium wurde alle 72 Stunden ersetzt. Jeder Tag über 5 Tage, zwei Platten für jeden der vier Induktionen wurden mit DMEM gewaschen tote Zellen zu entfernen. Lebende Zellen wurden mit Trypsin behandelt und Proben von jeder Platte wurden dreimal gezählt unter Verwendung eines Coulter Zellzähler (Coulter Corporation, Miami, FL, USA). Die durchschnittliche Anzahl von Zellen aus jeder Platte wurde berechnet, um die Wachstumsrate für jede Induktion zu bestimmen.
DNA Histogrammanalyse
Die Zellen wurden bei ungefähr 200 000 Zellen / 100 mm-Platte ausgesät und induzierte weder Protein, mutanten p53 allein, Wildtyp-p53 allein zu exprimieren oder beiden Proteine gleichzeitig. 3 Tage nach Induktion, beide schwimmenden Zellen in dem Medium und lebenden Zellen auf der Platte wurden mit 100% Ethanol und fixierten für mindestens 1 h bei 4 ° C. Die fixierten Zellen wurden mit 20 g / ml RNase A bei einer Konzentration von 2 Mio Zellen / ml in PBS 1 zentrifugiert und erneut suspendiert und 50 g / ml Propidiumiodid (Sigma Chemical Co. St. Louis, MO, USA). (; Becton Dickson, Menlo Park, CA, USA FACSCaliber) innerhalb von 4 h Die gefärbten Zellen wurden in einem Fluoreszenz-aktivierten Zellsortierer analysiert.
RNA-Isolierung und Northern-Blot-Analyse
Die Zellen wurden bei etwa 60% Konfluenz ausgesät und induzierte weder Protein, mutanten p53 allein, Wildtyp-p53 allein zu exprimieren oder beiden Proteine gleichzeitig für 16 Stunden. Die Zellen wurden durch Abkratzen und Kerne wurden auf Eis in hypotonischem Puffer A (10 mM HEPES, 1,5 m M MgCl2. 10 m M KCl und Protease-Inhibitor-Cocktail) extrahiert gesammelt für 10 min, gefolgt von 0,5% NP-40. Genomische DNA und Protein wurden für 10 min bei Raumtemperatur mit 1% Formaldehyd vernetzt. Die Proben wurden mit Ultraschall behandelt, 200- bis 1000-bp-DNA-Fragment zu ergeben. Insgesamt 1 / 50stel der Probe wurde für die Eingabe entfernt. Nach der Klärung durch Zentrifugation wurde beschallt Lysaten mit gleichen Volumen von 2 RIPA-Puffer (100 mM Tris-HCl pH 7,4, 2% NP-40, 0,5% Na-Desoxycholat, 300 m M NaCl, 2 mM EDTA) und 1 verdünnt Inhibitor-Cocktail. 100-Protease und immunpräzipitiert mit Anti-Flag-Antikörper M2, p53Pab 1801 / Pab 421 oder Myc 9E10 als negative Kontrolle. Die Immunkomplexe wurden durch Protein A / G Sepharose-Kügelchen eingefangen, die mit Lachssperma-DNA und nacheinander mit TSE-Puffer (0,1% SDS, 1% Triton X-100, 2 m M EDTA, 20 m M Tris-HCl pH 8,1) gesättigt waren enthaltend 150 m m NaCl, TSE, die 500 m m NaCl, Puffer III (0,25 m LiCl, 1% NP-40, 1% Natriumdesoxycholat, 1 m m EDTA, 10 m m Tris-HCl pH 8,1) und TE-Puffer ( 10 m m Tris-HCl pH 8,0, 0,1 m m EDTA). Die Immunkomplexe wurden dann zweimal mit Elutionspuffer (1% SDS, 0,1 M NaHCO & sub3;) für 15 min bei Raumtemperatur eluiert. Crosslinking wurde für mindestens 6 h bei 65 ° C aufgelöst. DNA wurde durch Phenol-Chloroform-Extraktion und Ethanolpräzipitation gereinigt und in 50 l TE resuspendiert. PCR wurde durchgeführt, das p21-Promotor-Fragment mit der 5'-Primer P2151 (CAGGCTGTGGCTCTGATTGG) und die 3'-Primer p2131 (TCCAGAGTAACAGGCTAAGG) zu erfassen. PCR wurde durchgeführt, die MDM2 genomische DNA mit dem 5'-Primer MDM2RE1F (GGGAGTTCAGGGTAAAGGTCA) und der 3'-Primer MDM2RE1R (CCTTTTACTGCAGTTTCG) zu erfassen. PCR wurde durchgeführt, das PIG3 Promotorfragment mit der 5'-Primer PIG3 RE F (CCAACGGCTCCTTTCTCTTC) und der 3'-Primer PIG3 RE R (GCTTGACAGAAAGTGCGATTC) zu erfassen.
Immunofluoreszenz
Referenzen
Anerkennungen
Diese Arbeit wird teilweise durch NIH Grant-2 RO1 CA076069 unterstützt.